OpenFold を使用してタンパク質の立体構造予測をやってみた
この記事は公開されてから1年以上経過しています。情報が古い可能性がありますので、ご注意ください。
2024 年のノーベル科学賞は AlphaFold を開発した Google DeepMind の研究者(開発者)でしたね。ノーベル化学賞受賞記念としてタンパク質の立体構造予測をやってみました。
はじめに
タンパク質の立体構造を予測できる OpenFold を AWS 上で動かしてみました。
AWS に不慣れな方でもはじめられるように GPU インスタンスと Docker を使用したシンプルな実行環境を構築します。
本記事では、以下の内容を説明します。
- OpenFold の実行環境構築手順
- 必要なデータベースのダウンロード方法
- 実際の予測実行と結果確認方法
Icons made by Freepik from Flaticon
OpenFoldとは
OpenFold は、タンパク質の立体構造を予測する機械学習モデルです。
Google DeepMind が開発した AlphaFold をベースにしたオープンソース実装です。
実行環境の準備
本記事では 2 種類のインスタンスを使用します。
準備作業用のインスタンス
時間のかかるデータベースのダウンロードと圧縮されたデータ展開は非 GPU インスタンスを利用しました。当初 T 系のインスタンスタイプを使用したのですが、早々にバーストクレジットが枯渇したため M 系のインスタンスタイプに変更しました。
- インスタンスタイプ:m7i-flex.large
- OS:Ubuntu 24.04 LTS
推論用 GPU インスタンス
OpenFold を実行(推論)するインスタンスは GPU インスタンスを利用します。OpenFold は現在 CUDA 11 と 12 をサポートしています。CUDA 12 の実行環境がセットアップ済みの AMI を利用しました。
- インスタンスタイプ:g6.xlarge(NVIDIA L4 Tensor Core GPU×1 搭載)
- OS:Ubuntu 22.04 LTS
- CUDA Version:12.4
- AMI:Deep Learning Base OSS Nvidia Driver GPU AMI (Ubuntu 22.04) 20241213(ami-0472e369a78a2dd1c)
$ nvidia-smi
Tue Dec 24 05:18:02 2024
+-----------------------------------------------------------------------------------------+
| NVIDIA-SMI 550.127.05 Driver Version: 550.127.05 CUDA Version: 12.4 |
|-----------------------------------------+------------------------+----------------------+
| GPU Name Persistence-M | Bus-Id Disp.A | Volatile Uncorr. ECC |
| Fan Temp Perf Pwr:Usage/Cap | Memory-Usage | GPU-Util Compute M. |
| | | MIG M. |
|=========================================+========================+======================|
| 0 NVIDIA L4 On | 00000000:31:00.0 Off | 0 |
| N/A 23C P8 11W / 72W | 1MiB / 23034MiB | 0% Default |
| | | N/A |
+-----------------------------------------+------------------------+----------------------+
+-----------------------------------------------------------------------------------------+
| Processes: |
| GPU GI CI PID Type Process name GPU Memory |
| ID ID Usage |
|=========================================================================================|
| No running processes found |
+-----------------------------------------------------------------------------------------+
ストレージ
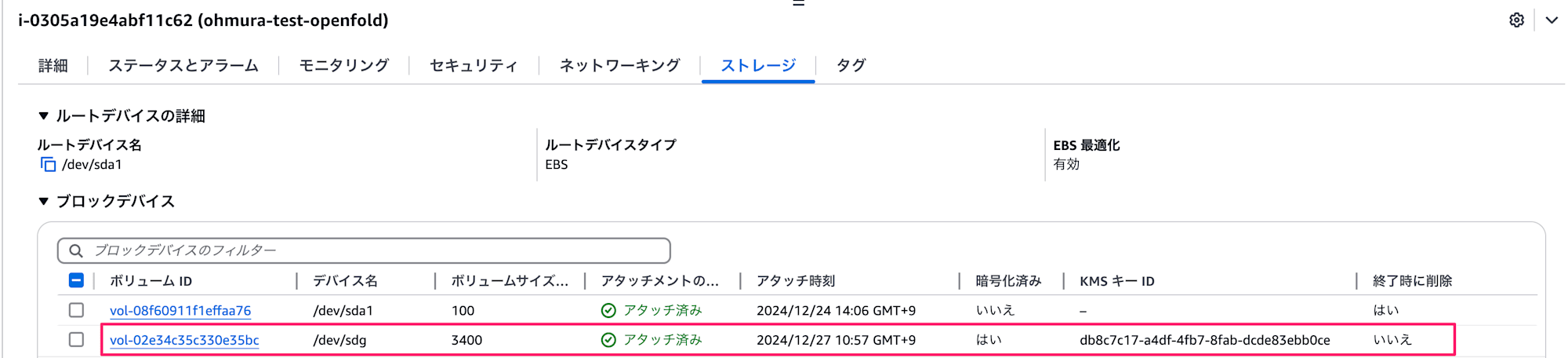
OpenFold の実行に必要なデータベースのサイズは 2.7TB の容量が必要です。今回は gp3 タイプの EBS ボリューム 3.4TB を追加して対応しました。データをダウンロード後、GPU インスタンスへ EBS をアタッチし直します。
$ df -h
Filesystem Size Used Avail Use% Mounted onk
/dev/nvme1n1 3.3T 2.8T 347G 90% /database
データベースの準備
OpenFold の実行には数種類のデータベースが必要です。これらをダウンロードするのにまず一苦労します。
OpenFold のリポジトリにデータベースファイルをダウンロード用のスクリプトが用意されています。
過去に何度かダウンロードしたことがあるのですが、ダウンロードの中断が発生してなかなかスムーズにダウンロードができません。
データベースのファイルが tar などで固めた上で圧縮している提供元がないかと探したらありました。AWS が EKS で OpenFold の環境構築用のサンプルアーキテクチャを公開しています。その中でデータベースのダウンロード用に S3 から圧縮されたファイル形式で提供されていました。今回はデータベースのダウンロードをスムーズに済ませるために、ダウンロード元はこちらを利用しました。
ダウンロードスクリプトの実行
ダウンロード用に用意した非 GPU インスタンスを利用します。
AWS S3 からデータベースをダウンロードするために、以下のスクリプトを実行します。3.4TB の EBS は/mnt/ebsにマウントしています。
cd /mnt/ebs
git clone https://github.com/aws-samples/aws-do-openfold-inference.git
cd /mnt/ebs/aws-do-openfold-inference/download-openfold-data
sudo apt install aria2 -y
scriptsディレクトリのあるパスまで移動して並列でダウンロードを走らせます。BFD のデータが一番最後に終わりました。ダウンロード自体は長くないのですが、ファイルの展開に数時間かかりました。合計すると一晩放置して待つことになりました。
nohup bash scripts/download_alphafold_params_s3.sh /mnt/ebs/openfold/data/ > 1.log &
nohup bash scripts/download_mgnify_s3.sh /mnt/ebs/openfold/data/ > 2.log &
nohup bash scripts/download_pdb_mmcif_s3.sh /mnt/ebs/openfold/data/ > 3.log &
nohup bash scripts/download_small_bfd_s3.sh /mnt/ebs/openfold/data/ > 4.log &
nohup bash scripts/download_uniprot_s3.sh /mnt/ebs/openfold/data/ > 5.log &
nohup bash scripts/download_bfd_s3.sh /mnt/ebs/openfold/data/ > 6.log &
nohup bash scripts/download_pdb70_s3.sh /mnt/ebs/openfold/data/ > 7.log &
nohup bash scripts/download_pdb_seqres_s3.sh /mnt/ebs/openfold/data/ > 8.log &
nohup bash scripts/download_uniclust30_s3.sh /mnt/ebs/openfold/data/ > 9.log &
nohup bash scripts/download_uniref90_s3.sh /mnt/ebs/openfold/data/ > 10.log &
OpenFold 実行時に必要だとわかった追加ファイル
OpenFold のパラメーターファイル
OpenFold 実行時に必要なパラメーターファイルがありませんでした。追加で必要なパラメーターファイルは以下のコマンドでダウンロードします。このときは GPU インスタンスに切り替えているため、3.4TB の EBS は/databaseにマウントしています。
DOWNLOAD_DIR="/database/openfold/data"
mkdir -p "${DOWNLOAD_DIR}"
aws s3 cp --no-sign-request --region us-east-1 s3://openfold/openfold_params/ "${DOWNLOAD_DIR}" --recursive
UnicClust30 追加ファイル
UniClust30 のデータベースはダウンロード済みです。ですが、必要なファイル(uniclust30_2018_08/uniclust30_2018_08)が足りていませんでした。OpenFold の公式な手順で必要なデータベースをダウンロードしました。新たにダウンロードしたファイル類は、既存のuniclust30ディレクトリ内に移動すれば問題なく OpenFold 動作しました。
[準備] 必要なソフトウェアのインストール
OpenFold を実行する GPU インスタンスに必要なソフトウェアをインストールします。
Docker のインストール
以下のコマンドで Docker をインストールします。
# Add Docker's official GPG key
sudo apt-get update
sudo apt-get install ca-certificates curl
sudo install -m 0755 -d /etc/apt/keyrings
sudo curl -fsSL https://download.docker.com/linux/ubuntu/gpg -o /etc/apt/keyrings/docker.asc
sudo chmod a+r /etc/apt/keyrings/docker.asc
# Add the repository to Apt sources
echo \
"deb [arch=$(dpkg --print-architecture) signed-by=/etc/apt/keyrings/docker.asc] https://download.docker.com/linux/ubuntu \
$(. /etc/os-release && echo "$VERSION_CODENAME") stable" | \
sudo tee /etc/apt/sources.list.d/docker.list > /dev/null
sudo apt-get update
sudo apt-get install docker-ce docker-ce-cli containerd.io docker-buildx-plugin docker-compose-plugin -y
OpenFold の準備
OpenFold のリポジトリをクローンし、CUDA 12 対応のブランチ(pl_upgrades)にチェックアウトします。
cd ~
git clone -b pl_upgrades https://github.com/aqlaboratory/openfold.git
Docker イメージのビルド
クローンしたリポジトリ内に Dockerfile が用意されています。そのままではビルドが通らなかったため修正が必要でした。
cd openfold
docker build -t openfold .
ビルド時のエラーメッセージから修正したのが以下のファイルです。バージョンを明示的に指定しました。
name: openfold-env
channels:
- conda-forge
- bioconda
- pytorch
- nvidia
dependencies:
- cuda
- gcc=12.4
- - python=3.10
+ - python=3.9.x
+ - numpy=1.22.4
- libgcc=7.2
- setuptools=59.5.0
- pip
- openmm=7.7
- pdbfixer
- pytorch-lightning
- biopython
- numpy<2.0.0
- pandas
- PyYAML==5.4.1
- requests
- scipy
- tqdm==4.62.2
- typing-extensions
- wandb
- modelcif==0.7
- awscli
- ml-collections
- mkl=2022.1
- aria2
- git
- bioconda::hmmer
- bioconda::hhsuite
- bioconda::kalign2
- pytorch::pytorch=2.1
- pytorch::pytorch-cuda=12.1
- pip:
- deepspeed==0.12.4
- dm-tree==0.1.6
- git+https://github.com/NVIDIA/dllogger.git
- flash-attn
これで OpenFold の Docker イメージが準備できました。
OpenFold 実行
OpenFold のリポジトリ内にテストデータとして、6KWC のアミノ酸配列が用意されています。こちらのアミノ酸配列を使ってタンパク質の立体構造予測を試してみます。
テスト実行
以下のスクリプトを使用して OpenFold を実行します。別のインスタンスでダウンロードしたデータベースが格納されている EBS は/databaseにマウントしています。
#!/bin/sh
cd /home/ubuntu/openfold/examples/monomer
docker run \
--gpus all \
-v $PWD/:/data \
-v /database/openfold/data/:/database \
-ti openfold:latest \
python3 /opt/openfold/run_pretrained_openfold.py \
/data/fasta_dir \
/database/pdb_mmcif/mmcif_files/ \
--uniref90_database_path /database/uniref90/uniref90.fasta \
--mgnify_database_path /database/mgnify/mgy_clusters_2022_05.fa \
--pdb70_database_path /database/pdb70/pdb70 \
--uniclust30_database_path /database/uniclust30/uniclust30_2018_08/uniclust30_2018_08 \
--bfd_database_path /database/bfd/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt \
--jackhmmer_binary_path /opt/conda/bin/jackhmmer \
--hhblits_binary_path /opt/conda/bin/hhblits \
--hhsearch_binary_path /opt/conda/bin/hhsearch \
--kalign_binary_path /opt/conda/bin/kalign \
--openfold_checkpoint_path /database/openfold_params/finetuning_ptm_2.pt \
--config_preset "model_1_ptm" \
--model_device "cuda:0" \
--output_dir /data/results
==========
== CUDA ==
==========
CUDA Version 12.1.1
Container image Copyright (c) 2016-2023, NVIDIA CORPORATION & AFFILIATES. All rights reserved.
This container image and its contents are governed by the NVIDIA Deep Learning Container License.
By pulling and using the container, you accept the terms and conditions of this license:
https://developer.nvidia.com/ngc/nvidia-deep-learning-container-license
A copy of this license is made available in this container at /NGC-DL-CONTAINER-LICENSE for your convenience.
[2024-12-27 03:17:59,782] [INFO] [real_accelerator.py:161:get_accelerator] Setting ds_accelerator to cuda (auto detect)
INFO:/opt/openfold/openfold/utils/script_utils.py:Loaded OpenFold parameters at /database/openfold_params/finetuning_ptm_2.pt...
INFO:/opt/openfold/run_pretrained_openfold.py:Generating alignments for 6KWC_1...
参考: Original OpenFold README - OpenFold documentation
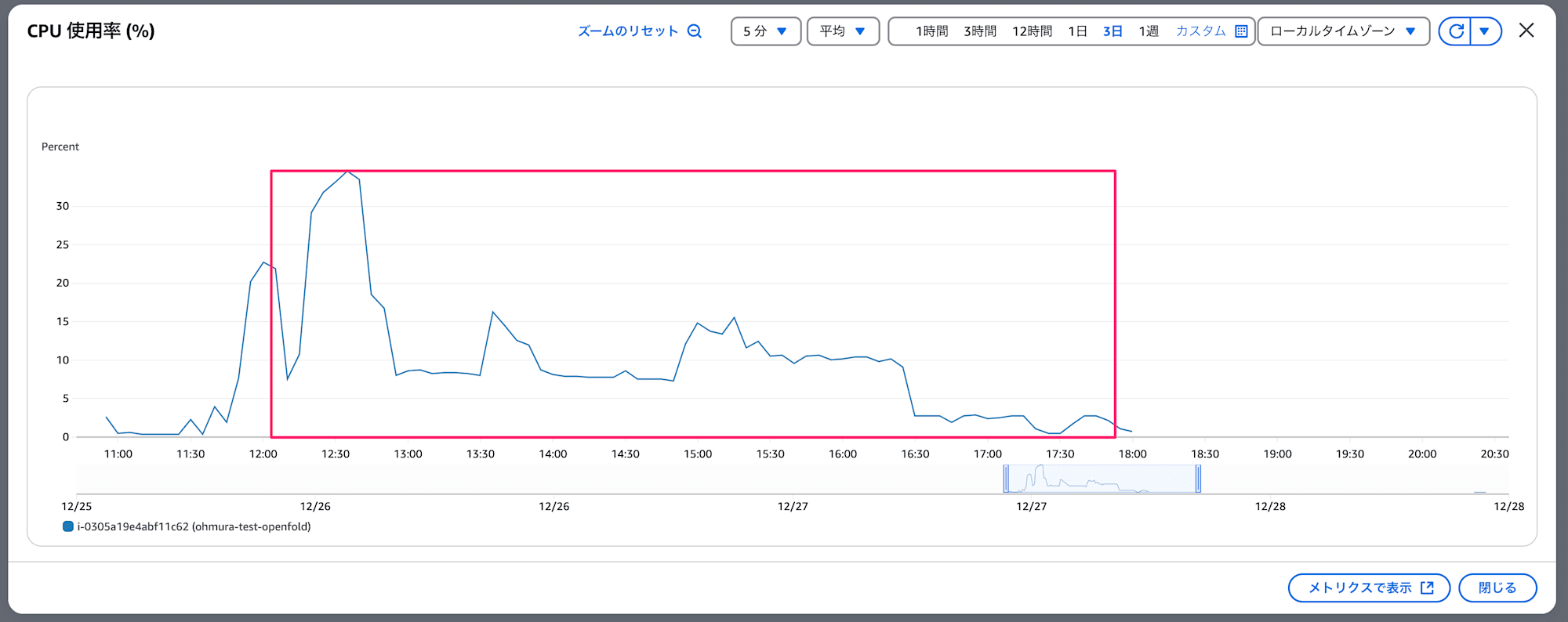
推論に 6 時間かかりました。GPU が 1 台というよりも、メモリがあればもう少し早く終わったのではないかなと思います。キャプチャは推論していた時間帯の CPU 使用率の推移です。
実行結果確認
実行結果は--output_dirで指定したディレクトリに保存されます。出力サイズは 6.7MB でした。6KWC_1_model_1_ptm_relaxed.pdbを可視化ツールで開いて結果を確認してみます。
$ tree .
.
├── alignments
│ └── 6KWC_1
│ ├── bfd_uniclust_hits.a3m
│ ├── hhsearch_output.hhr
│ ├── mgnify_hits.sto
│ └── uniref90_hits.sto
├── predictions
│ ├── 6KWC_1_model_1_ptm_relaxed.pdb
│ ├── 6KWC_1_model_1_ptm_unrelaxed.pdb
│ └── timings.json
└── timings.json
参考: https://openfold.readthedocs.io/en/latest/Inference.html#model-outputs
可視化してみる
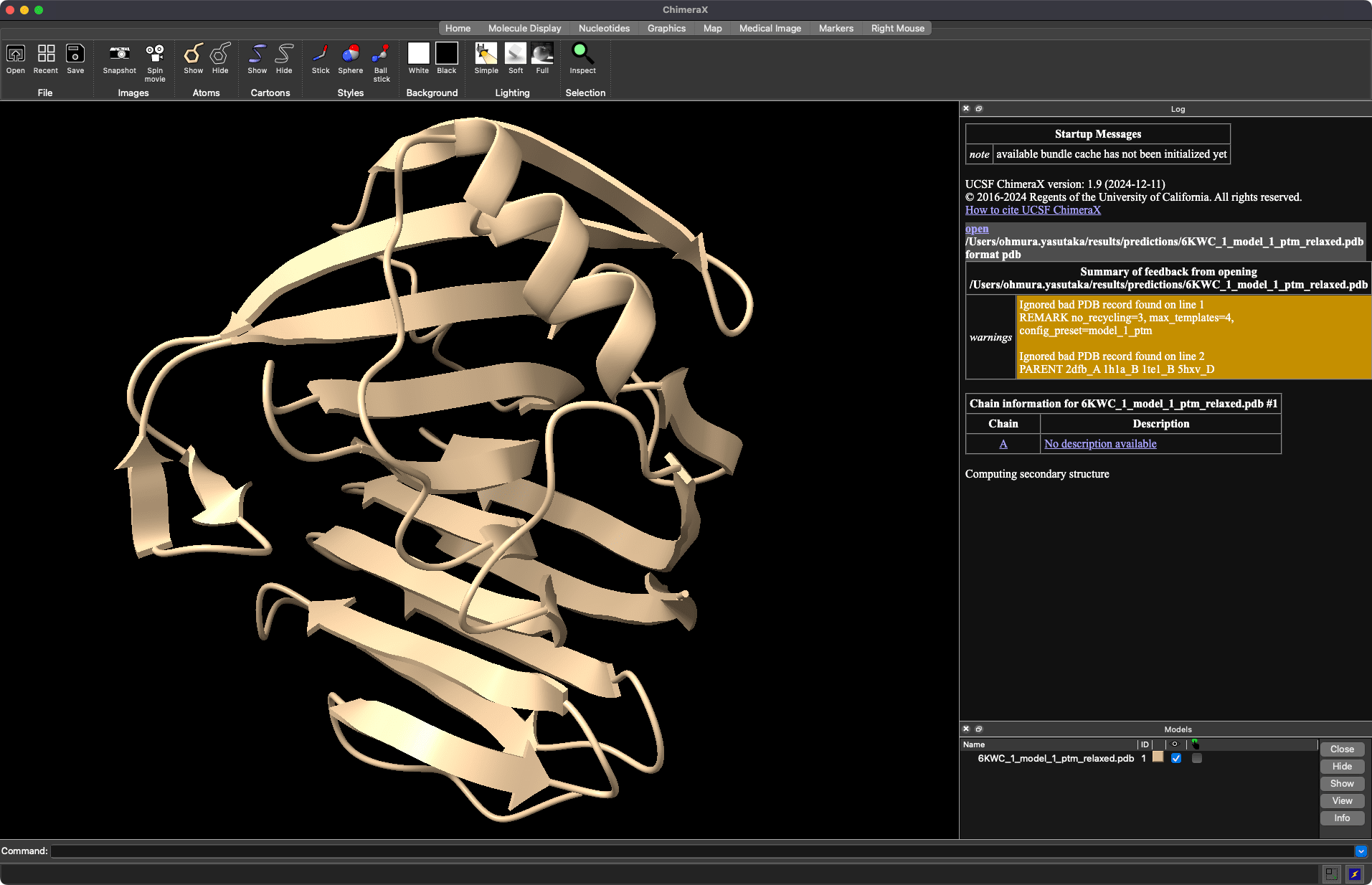
可視化ツールは、UCSF ChimeraX を利用します。Mac ですと M1(Arm)チップにも対応しています。
ChimeraX で開いてみた
正常に読み込めました。
raibowコマンドで着色しました。いい感じですね、よく見かけるイメージになりました。
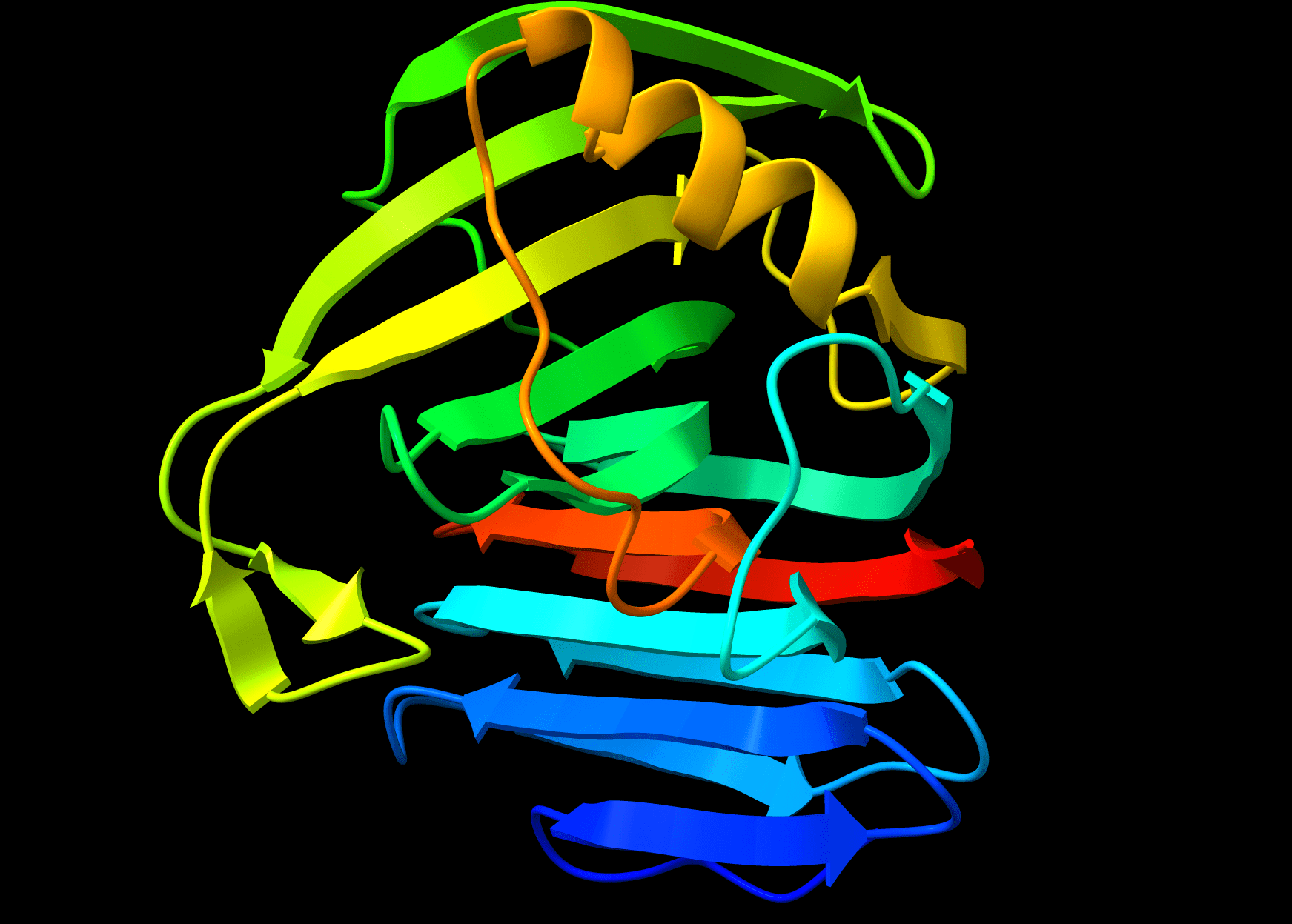
参考: rainbow
3D モデルなのでグリグリ動かすことができます。
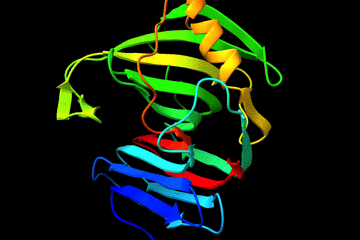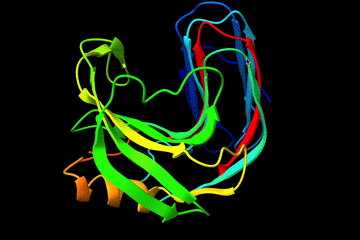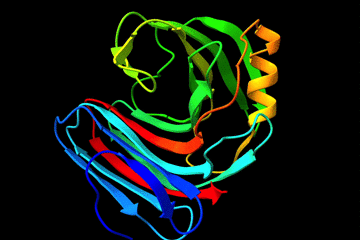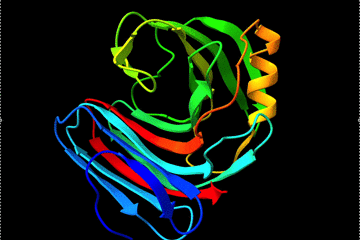
X 線結晶構造解析の結果と見比べてみる
タンパク質の立体構造を調べるために従来から使われている手法として、X 線結晶構造解析があります。
X線結晶構造解析は、創薬で最も使われている手法です。この手法では、タンパク質を結晶化した後に、X線を照射し、その回折像から構造解析を行います。結晶化が可能なタンパク質のみに適用可能な手法ですが、分子量の制限が無いことや、高いスループット性など他の手法より優れた点があります。
2. X線結晶構造解析の利点 | AgroBox アグロデザイン・スタジオ
今回、タンパク質の立体構造予測したアミノ酸配列 6KWC の X 線結晶構造解析した結果と見比べてみましょう。PDB(Protein Data Bank)にデータがアップされています。めちゃめちゃ似ていますね、予測の精度は良さそうです。
まとめ
Docker コンテナで環境さえ作ってしまえば動くので手軽にはじめられることがわかりました。
- OpenFold を GPU インスタンス 1 台だけのシンプルな構成で実行しました
- Docker コンテナを使用することで、環境構築の手間を削減できました
- 約 3TB のデータベースの準備が必要なため、初回の実行環境セットアップには時間がかかります
おわりに
ノーベル化学賞受賞記念として「タンパク質の立体構造予測をやってみた」ブログを 2024 年中に投稿できて悔いのない年となりました。推論完了後に GPU インスタンスを自動停止したいとなると、AWS ParallelCluster, AWS Batch, AWS PCS このあたりのサービスで OpenFold を動かすと相性が良さそうです。
研究されている方なら共用のスパコンなどでセットアップ済みの環境がすでにあるかと思います。昨今の GPU 需要で GPU 環境で実行できるまでのキューの待ち時間が長いようでしたら、何かしらのクラウドサービスで急ぎ実行可能な環境あっても良いのではないでしょうか、Dokcer コンテナさえあれば動きましたので。身近な IT 詳しい方に相談してみてください。
最後に 2025 年は OpenFold 3 の登場をお待ちしております。